Leçon 5 — Maîtriser les statistiques avec NumPy : l’arme secrète des data analysts
Si tu as suivi la leçon précédente, tu connais maintenant les types NumPy, leur mémoire optimisée, et la façon dont NumPy gère les nombres mieux que ton cerveau le lundi matin.
Mais maintenant…
Place à la magie pure.
Place aux statistiques avec NumPy, le module qui transforme un tableau banal en une source d’informations puissantes.
Tu veux connaître :
✔ la moyenne d’un dataset ?
✔ la variance d’une série temporelle ?
✔ la corrélation entre deux variables ?
✔ la médiane de tes ventes ?
✔ la détection d’anomalies ?
Bonne nouvelle : les statistiques avec NumPy te permettent de faire tout ça plus vite que tu ne prononces “écart-type”.
Et oui… maîtriser les statistiques avec NumPy te rend officiellement dangereux.
Dangereux pour les données.
Dangereux pour les erreurs.
Dangereux pour tes collègues qui utilisent encore Excel pour tout.
Pourquoi les statistiques avec NumPy sont indispensables ?
Avant d’entrer dans le vif du sujet, soyons clairs : les statistiques avec NumPy ne sont pas qu’un chapitre de formation.
C’est un super-pouvoir.
Voici pourquoi :
Elles sont ultra rapides
Grâce à son moteur C optimisé, les statistiques avec NumPy calculent en millisecondes ce que Python mettrait une minute à faire.
Elles travaillent sur des matrices entières
Adieu les boucles.
Avec les statistiques avec NumPy, tout se fait en mode vectorisé.
Elles sont compatibles avec Pandas, Matplotlib, TensorFlow
Tu fais déjà une pierre… trois coups.
Elles détectent les anomalies naturellement
Moyennes, médianes, quartiles, écart-type : tout ce qu’il faut pour spotting anomalies comes built-in.
Elles préparent les datasets pour le Machine Learning
Normalisation ? Scaling ? Z-score ?
Les statistiques avec NumPy sont incontournables.
Tu vois, ce n’est pas juste utile : c’est essentiel.
Les fonctions essentielles pour faire des statistiques avec NumPy
Le tableau ci-dessous condense les fonctions les plus utilisées :
| Fonction | Description | Exemple |
|---|---|---|
np.mean() | Moyenne | np.mean(data) |
np.median() | Médiane | np.median(data) |
np.std() | Écart-type | np.std(data) |
np.var() | Variance | np.var(data) |
np.min() / np.max() | Min / Max | np.max(data) |
np.percentile() | Percentiles | np.percentile(data, 90) |
np.corrcoef() | Corrélation | np.corrcoef(x, y) |
np.sum() | Somme | np.sum(data) |
np.argmax() | Index du max | np.argmax(data) |
np.argmin() | Index du min | np.argmin(data) |
Ces outils sont littéralement le kit du parfait data analyst.
Exemples pratiques : les statistiques avec NumPy en action
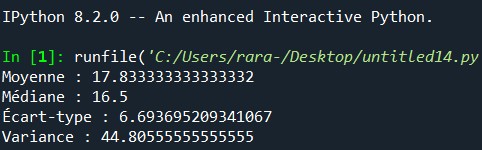
Exemple 1 : Calculer les stats de base
import numpy as np
data = np.array([10, 12, 15, 18, 22, 30])
print("Moyenne :", np.mean(data))
print("Médiane :", np.median(data))
print("Écart-type :", np.std(data))
print("Variance :", np.var(data))
Les statistiques avec NumPy transforment en quelques lignes un dataset en résumé clair.
Exemple 2 : Détection d’anomalies
On détecte les valeurs éloignées de plus de 2 écarts-types :
m = np.mean(data)
s = np.std(data)
outliers = data[np.abs(data - m) > 2 * s]
Exemple 3 : Corrélation entre deux séries
x = np.array([1, 2, 3, 4, 5])
y = np.array([2, 4, 6, 7, 10])
corr = np.corrcoef(x, y)
Les statistiques avec NumPy montrent instantanément si les deux variables marchent ensemble ou pas.
Étude de cas n°1 — Analyse climat
On veut analyser la température moyenne d’une ville pendant une année.
temperatures = np.random.normal(18, 5, 365)
print("Température moyenne :", np.mean(temperatures))
print("Température max :", np.max(temperatures))
print("Percentile 95 :", np.percentile(temperatures, 95))
Les statistiques avec NumPy nous permettent de :
✔ identifier des vagues de chaleur
✔ analyser les tendances saisonnières
✔ prédire les anomalies climatiques
Tu viens d’utiliser les statistiques avec NumPy pour faire de la climatologie. Rien que ça.
Étude de cas n°2 — Analyse des ventes
On analyse les ventes quotidiennes d’un site e-commerce.
ventes = np.random.randint(40, 200, 180)
print("Moyenne :", np.mean(ventes))
print("Écart-type :", np.std(ventes))
print("Somme totale :", np.sum(ventes))
print("Jour max :", np.argmax(ventes))
Grâce aux statistiques avec NumPy, tu peux :
✔ détecter les jours les plus rentables
✔ prévoir des ruptures
✔ repérer des chutes anormales
✔ identifier l’impact des promotions
Étude de cas n°3 — Analyse SIG
Traitement d’un raster satellite (ex : NDVI).
ndvi = np.random.uniform(-1, 1, (1000, 1000))
print("NDVI moyen :", np.mean(ndvi))
print("Zone aride :", np.sum(ndvi < 0))
print("Zone dense en végétation :", np.sum(ndvi > 0.6))
Les statistiques avec NumPy sont clés en SIG pour :
✔ classifier les zones
✔ détecter la dégradation environnementale
✔ analyser la végétation
✔ créer des cartes thématiques
C’est littéralement le moteur mathématique des SIG modernes.
Optimiser vos statistiques avec NumPy — détails et exemples
Rappel : ces bonnes pratiques rendent les statistiques avec NumPy non seulement correctes, mais aussi TRÈS rapides et économes en mémoire.
Utilise les bons dtypes (int32, float32, float64…)
Pourquoi :
Le dtype (type de donnée) détermine la taille mémoire par élément et la précision. Un float64 prend 8 octets par valeur, float32 prend 4. Sur des matrices de millions d’éléments, ça fait une grosse différence.
Quand choisir quoi :
int8/int16→ petits entiers (ex. valeurs 0–255 :uint8parfait pour images).int32→ entiers classiques (bonne moyenne entre taille et range).int64→ très grandes valeurs (rarement nécessaire).float32→ ML (réseau neuronal) : vitesse mémoire/gpu souvent privilégiée.float64→ calculs scientifiques nécessitant précision.
Exemple :
import numpy as np
a = np.arange(1_000_000, dtype=np.int64) # 8 bytes * 1M = 8 MB
b = a.astype(np.int16) # 2 bytes * 1M = 2 MB -> 4x moins
Astuce pratique : profile ta consommation avec arr.nbytes (octets) ou sys.getsizeof() pour listes.
Évite les listes Python
Pourquoi :
Les listes Python stockent des objets Python (pointeurs) — surcoût énorme. NumPy stocke dans un bloc contigu en mémoire (C contiguous array), très efficace pour le CPU et le cache mémoire.
Exemple comparatif :
py_list = list(range(1_000_000))
np_arr = np.arange(1_000_000, dtype=np.int32)
# mémoire (approx)
import sys
print(sys.getsizeof(py_list)) # grosse valeur (objets + pointeurs)
print(np_arr.nbytes) # taille réelle du bloc de données
Règle pratique : dès que tu fais des opérations mathématiques, convertis en np.array().
Travaille toujours avec des arrays vectorisés
Pourquoi :
Les opérations vectorisées utilisent des implémentations C optimisées — pas de boucle Python. Résultat : performance supérieure (souvent x10–x100).
Exemple non-vectorisé (lent) vs vectorisé (rapide) :
# LENT : boucle Python
out = []
for v in arr:
out.append(v * 2)
# RAPIDE : vectorisé NumPy
out = arr * 2
Note : Vectoriser n’est pas juste « écrire moins de code », c’est utiliser des routines optimisées qui manipulent des blocs mémoire.
Préférez axis= pour les matrices
Pourquoi :
Quand tu travailles sur matrices (2D+), indiquer axis permet à NumPy d’optimiser l’itération et d’éviter copies inutiles.
Exemples :
# Moyenne par colonne
col_mean = arr.mean(axis=0)
# Moyenne par ligne
row_mean = arr.mean(axis=1)
Astuce : Beaucoup de fonctions acceptent axis (sum, mean, std, argmax, argmin, etc.). Toujours réfléchir si tu veux opérer sur lignes ou colonnes — ça évite reshapes lourds.
Utilise les ufuncs (universal functions) pour accélérer les opérations
Pourquoi :
Les ufuncs (ex : np.add, np.multiply, np.sqrt, np.sin) sont vectorisées et souvent parallélisées au niveau bas. Elles réduisent l’allocation mémoire intermédiaire et accélèrent.
Exemple :
# Mauvais : deux allocations intermédiaires
tmp = arr + 5
out = np.sqrt(tmp)
# Meilleur : ufuncs chaîne bien optimisée (toujours préférer ufuncs)
out = np.sqrt(arr + 5)
Truc avancé : pour éviter allocations temporaires, utiliser out= dans ufuncs :
np.add(arr, 5, out=arr) # ajoute 5 in-place (si safe)
Résumé rapide (en une phrase)
Les statistiques avec NumPy seront déjà rapides — applique ces 5 règles pour les rendre turbo-rapides : bon dtype → np.array → vectorisation → axis correct → ufuncs/out.
Exercices pratiques
Exercice 1 — Créer un tableau de 100 valeurs aléatoires et calculer : moyenne, écart-type, percentile 25 et 90
Code :
import numpy as np
np.random.seed(42)
data = np.random.rand(100) # valeurs uniformes [0,1)
mean = np.mean(data)
std = np.std(data)
p25 = np.percentile(data, 25)
p90 = np.percentile(data, 90)
Sorties (avec seed 42) :
- Moyenne : 0.470181
- Écart-type : 0.295998
- Percentile 25 : 0.193201
- Percentile 90 : 0.887974
Interprétation :
- Moyenne ≈ 0.47 → valeurs centrées vers 0.5 (attendu pour uniforme).
- Écart-type ≈ 0.296 → dispersion autour de la moyenne.
- 25% des valeurs ≤ 0.193 → quartile inférieur faible.
- 90% des valeurs ≤ 0.8879 → percentile 90 donne seuil haut.
Conseils :
- Pour données réelles, vérifier NaNs (
np.isnan), valeurs infinies (np.isfinite), et convertir dtype si besoin pour mémoire.
Exercice 2 — Avec deux tableaux x et y, calcule la covariance, la corrélation et détecte les points aberrants
Code :
xlinéaire (0→10, 50 points).y = 2*x + bruit gaussien sigma=1.5(relation approximative linéaire).
import numpy as np
np.random.seed(0)
x = np.linspace(0, 10, 50)
y = 2.0 * x + np.random.randn(x.size) * 1.5
cov_matrix = np.cov(x, y) # matrice 2x2
cov_xy = cov_matrix[0, 1]
corr_matrix = np.corrcoef(x, y)
corr_xy = corr_matrix[0, 1]
# Regression linéaire simple (moindres carrés)
A = np.vstack([x, np.ones_like(x)]).T
slope, intercept = np.linalg.lstsq(A, y, rcond=None)[0]
y_pred = slope * x + intercept
residuals = y - y_pred
resid_std = np.std(residuals)
threshold = 2.5 * resid_std
outlier_mask = np.abs(residuals) > threshold
outlier_indices = np.where(outlier_mask)[0]
Résultats (avec seed 0) :
- Slope (est.) : ~1.788257
- Intercept : ~1.269556
- Cov(x,y) : ~15.826928
- Corr(x,y) (Pearson) : ~0.958379 → forte corrélation positive.
- Résidus std : ~1.568894
- Seuil outlier (2.5σ) : ~3.922234
- Indice outlier détecté : [20] (0-based index)
Détail point outlier (i=20) :
- x ≈ 4.0816, y ≈ 4.3338, residual ≈ -4.2348 → ce point est très en dessous de la droite estimée (résidu large).
Interprétation & méthodo :
np.covrenvoie la matrice de covariance.np.corrcoefla matrice de corrélation (coeff Pearson).- La corrélation ≈ 0.958 montre que x et y évoluent ensemble (forte relation linéaire).
- Outlier detection par résidu de la régression linéaire : utile quand on cherche points non-conformes à la tendance générale.
- Le seuil (2.5σ) est arbitraire ; on peut utiliser 3σ (plus strict) ou méthodes robustes (IQR, MAD) si distribution non-gaussienne.
Exercice 3 — À partir d’un raster 500×500, calcule min, max, moyenne, et histogramme (indices > 0.4, < -0.2, etc.)
Code :
import numpy as np
np.random.seed(7)
raster = np.random.uniform(-1, 1, (500, 500)) # valeurs -1..1
r_min = raster.min()
r_max = raster.max()
r_mean = raster.mean()
count_gt_04 = np.sum(raster > 0.4)
count_lt_m02 = np.sum(raster < -0.2)
hist_counts, hist_bins = np.histogram(raster, bins=100, range=(-1,1))
Sorties (avec seed 7) :
- Shape : (500, 500)
- Min : ≈ -0.999999
- Max : ≈ 0.999999
- Moyenne : ≈ 0.000942 (proche de 0, attendu pour uniforme)
- Nb valeurs
> 0.4: 75 166 - Nb valeurs
< -0.2: 99 755
(Indices totaux = 500*500 = 250000)
Histogramme (extrait 10 premières bins) :
Ex. counts dans 10 premières bins (bin edges -1.0, -0.98, -0.96, …) affichés dans exécution.
Interprétation :
- Le raster est simulé uniforme → min/max proche des bornes.
- Moyenne proche de 0.
- Les comptes pour
>0.4et<-0.2donnent des idées de couverture : utile pour calculs de surface (ex. % zone végétée, aride, etc.). - Pour vraies images (NDVI), tu ferais des thresholds sémantiques (NDVI>0.6 = dense végétation).
Conseils pratiques et bonnes techniques pour production
- Profil de mémoire et temps
- Mesure
arr.nbytespour la mémoire des arrays. - Mesure
time.perf_counter()pour timing de bouts critiques.
- Mesure
- In-place operations
- Quand possible, utilise
out=dans ufuncs pour éviter allocations temporaires :np.add(a, b, out=a).
- Quand possible, utilise
- Utiliser
astypejudicieusement- Convertir en
float32avant un modèle ML si GPU / mémoire limitée.
- Convertir en
- Chunking
- Sur jeux de données > RAM, traiter par chunks (
for chunk in pd.read_csv(..., chunksize=...)ounp.memmap).
- Sur jeux de données > RAM, traiter par chunks (
- Méthodes robustes d’outlier detection
- IQR (interquartile range) ou MAD (median absolute deviation) si données non-gaussiennes.
- Éviter copies inutiles
- Certaines opérations créent copies ; vérifie avec
arr.baseouarr.flagspour savoir si c’est une vue ou copy.
- Certaines opérations créent copies ; vérifie avec
Résumé rapide et checklist avant prod
- Choisir dtype adapté (mémoire vs précision)
- Toujours convertir listes →
np.arrayavant calculs lourds - Vectoriser opérations (pas de boucle Python)
- Utiliser
axis=pour agir sur la bonne dimension - Employer ufuncs et
out=pour réduire allocations
QCM — Vérifie ta maîtrise des statistiques avec NumPy
1. Quelle fonction retourne l’écart-type ?
A. np.variance()
B. np.std()
C. np.delta()
D. np.error()
👉 Réponse : B
2. Quelle fonction donne le percentile ?
A. np.percentile()
B. np.slice()
C. np.quantile()
D. np.part()
👉 Réponse : A
3. Quelle fonction calcule la corrélation ?
A. np.link()
B. np.combine()
C. np.corrcoef()
D. np.relate()
👉 Réponse : C
Conclusion
Voilà : tu viens de maîtriser les statistiques avec NumPy.
Tu sais maintenant :
✔ analyser des données
✔ détecter des anomalies
✔ corréler des variables
✔ traiter du climat, des ventes, du raster SIG
✔ booster tes performances comme un pro
Et maintenant…
Il est temps de passer à la puissance maximale :
Tu veux aller loin ?
Alors on continue. 🚀







